DNA -Replikationsmechanismen in Prokaryoten und Eukaryoten
- 2449
- 447
- Said Ganzmann
Der Replikation von DNA (Desoxyribonukleinsäure) Es besteht darin, das Genom zu kopieren, dh alle genetischen Informationen, die in der DNA eines Organismus enthalten sind, um zwei identische Kopien zu erzeugen. Das Genom verfügt über die notwendigen Informationen, um einen vollständigen Organismus aufzubauen.
Vor der Zellteilung tritt die DNA -Replikation auf. Durch Meiose treten Gameten für die sexuelle Reproduktion auf. Durch Mitose tritt der Zellersatz (zum Beispiel Haut und Blut) und die Entwicklung (zum Beispiel Gewebe und Organe) auf.
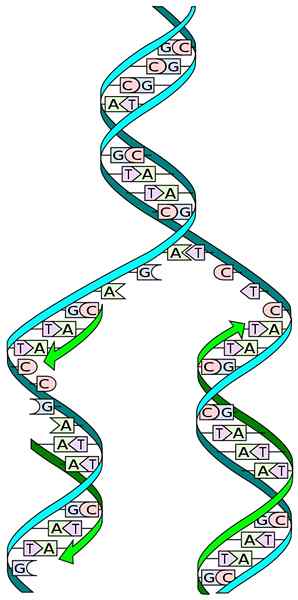
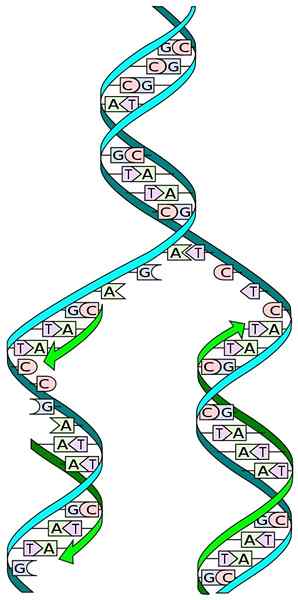 Quelle: I, MadPrime [CC0]
Quelle: I, MadPrime [CC0] Das Wissen der DNA -Struktur ermöglicht es uns zu verstehen, wie ihre Replikation auftritt. Die Struktur der DNA besteht aus einem Doppelpropeller, der aus zwei Antipaallalketten aufeinanderfolgender Nukleotide besteht, deren Stickstoffbasen spezifisch ergänzt werden.
Während der Replikation wirkt jede der Ketten der Doppel -DNA -Kette als Form für die Biosynthese einer neuen Kette. Die beiden neu synthetisierten Ketten haben Basen, die zu den Basen der Schimmelpilzkette komplementär sind: Adenin (a) mit Timina (T) und Cytosin (c) mit Guanin (G).
Mehrere Enzyme und Proteine nehmen an der DNA -Replikation teil. Zum Beispiel den Doppel-DNA-Propeller öffnen, die DNA offen halten und Desoxyribonukleosidos-5'-Triphosphat (DNTP) zur Bildung der neuen Kette hinzufügen.
[TOC]
Die DNA -Replikation ist semi -konservativ
Basierend auf der Struktur von DNA schlugen Watson und Crick vor, dass die DNA -Replikation semi konservativ auftritt. Dies wurde von Meselson und Stahl durch Markierung der DNA von demonstriert Escherichia coli Mit dem schweren Isotop von Stickstoff, fünfzehnN folgt für mehrere Generationen das Verteilungsmuster in einem Kulturmedium mit Lichtstickstoff, 14N.
Messelson und Stahl stellten fest, dass in der ersten Generation die beiden DNA -Moleküle der Töchter, jedes Molekül, das mit einer Kette mit dem schweren Stickstoffisotop und einem anderen mit dem leichten Isotop gekennzeichnet war. Im Gegensatz zum elterlichen DNA -Molekül, bei dem die beiden Ketten mit dem schweren Isotop gekennzeichnet waren, fünfzehnN.
In der zweiten Generation waren 50% der DNA -Moleküle wie die der ersten Generation, und die anderen 50% hatten nur leichten Stickstoff. Die Interpretation dieses Ergebnisses ist, dass der Propeller der Doppel -Tochter eine Elternkette (die als Form funktioniert) und eine neue Kette hat.
Der semi -konservative Replikationsmechanismus impliziert die Trennung von DNA -Ketten und die Paarung komplementärer Basen mittels aufeinanderfolgender Nukleotide, wodurch zwei Doppeltöchtertöchter erzeugt werden.
Replikation in Batterien
Beginn der DNA -Replikation in Bakterien
Die Bakterien -DNA besteht aus einem kreisförmigen Chromosom und hat nur einen Ursprungsort der Replikation. Von dieser Stelle an. Am Ende sind die Gabeln, um die Replikation abzuschließen.
Die Replikation beginnt mit der Vereinigung von DNAA -Proteinen zur Herkunftsstelle. Diese Proteine bilden wiederum einen Komplex. Dann sind die Hu- und IHF -Proteine gebunden.
Als nächstes werden DNAC -Proteine verbunden, mit denen die Helikasen der DNA verbindet. Diese helfen dabei. Die beiden Ketten trennen also noch mehr und bilden zwei einfache Ketten.
Kann Ihnen dienen: Was ist die chromosomale Permutation?Topoisomerase II oder DNA Girasa bewegt sich vor der DNA Helicy. SSB -Proteine (einzelne Ketten -DNA -Bindung) halten die DNA -Ketten getrennt. So kann die Biosynthese der Tochterkette beginnen.
Biosynthese von Töchtern DNA -Ketten in Bakterien
Das Prima -Enzym ist für die Synthese kurzer RNA -Ketten als Primer verantwortlich, die 10 bis 15 Nukleotide haben. Die DNA.
Da DNA -Ketten antiparallel sind, wird in der Führungskette ein Primer synthetisiert und viele Primer in der verzögerten Kette. Aus diesem Grund ist die Biosynthese der verzögerten Kette diskontinuierlich. Obwohl DNA -Ketten antiparallel sind, bewegt sich die Replikationsgabel in eine Richtung.
Die DNA -Polymerase ist für die Bildung kovalenter Bindungen zwischen benachbarten Nukleotiden der neu synthetisierten Ketten in 5'®3 'Richtung verantwortlich. In UND. coli, Es gibt fünf Polymerase -DNA: DNA -Polymerasen I und III führen eine DNA -Replikation durch; und die DNA -Polymerasen II, IV und V sind für die Replikation und Replikation der beschädigten DNA verantwortlich.
Der größte Teil der Replikation erfolgt durch DNA -Polymerase III, ein Holoenzym mit 10 verschiedenen Untereinheiten mit mehreren Funktionen bei der Replikation von DNA. Zum Beispiel ist die Alpha -Untereinheit für die Herstellung von Bindungen zwischen den Nukleotiden verantwortlich.
Ein Enzymkomplex ist für die Replikation von DNA in Bakterien verantwortlich
Die Helikase von DNA und Prima bildet einen Komplex namens Primosoma. Dies bewegt sich entlang der DNA und wirkt koordiniert, um die beiden Elternketten zu trennen und die Primer jedes bestimmte Intervall auf der verzögerten Kette zu synthetisieren.
Primosom bindet physisch an die DNA -Polymerase III und bildet das Replisom. Zwei DNA -Polymerasen III sind für die Replikation der DNA der Führung und der verzögerten Ketten verantwortlich. In Bezug auf die DNA -Polymerase III bildet die verzögerte Kette eine Schleife aus, die es ermöglicht, Nukleotide zu dieser Kette in derselben Richtung der Führungskette aufzurufen.
Die Zugabe von Nukleotiden zur Führungskette ist kontinuierlich. Während in der Verzögerung diskontinuierlich ist. Es werden Fragmente von 150 Längennukleotiden gebildet, die als Okazaki -Fragmente bezeichnet werden.
Die Exonuklease -Aktivität 5 ' -> 3' der DNA -Polymerase I ist für die Eliminierung der Primer und das Füllen verantwortlich, indem sie Nukleotide hinzufügen. Eine Enzymligase versiegelt die Lücken zwischen Fragmenten. Die Replikation endet, wenn sich die beiden Replikation in einer Abschlusssequenz befinden.
Das Protein, das Sie an die Terminationssequenz bindet, stoppt die Bewegung der Replikationsgabel. Topoisomerase II ermöglicht die Trennung der beiden Chromosomen.
Desoxiribonukleotide Tryphosphate werden durch DNA -Polymerase verwendet
Dexinukleosid -Tryphosphat (DNTP) enthält drei Phosphatgruppen, die mit Kohlenstoff 5 'von Desoxyribosa vereint sind. Die DNTPS (DATP, DTTP, DGTP und DCTP) verbinden die Formkette nach der AT/GC -Regel.
Kann Ihnen dienen: PlesiomorphieDie DNA-Polymerase katalysiert die folgende Reaktion: Die Hydroxylgruppe (-OH) 3 'des Nucleotids der Wachstumskette reagiert mit dem Alpha-Phosphat des eingehenden DNTP und setzt anorganische Pyrophosphat (PPI) (PPI) frei (PPI). Die PPI -Hydrolyse erzeugt Energie für die Bildung der kovalenten Bindung oder Phosphodiéster -Bindung zwischen den Nukleotiden der wachsenden Kette.
Mechanismen, die die Replikationstreue von DNA gewährleisten
Während der DNA -Replikation macht DNA -Polymerase III einen Fehler pro 100 Millionen Nukleotide. Obwohl die Fehlerwahrscheinlichkeit sehr niedrig ist, gibt es Mechanismen, die die Treue in der DNA -Replikation sicherstellen. Diese Mechanismen sind:
1) Stabilität im Basisapar. Die Wasserstoffbrückenbindungsenergie zwischen AT/GC ist größer als in fehlerhaften Basenpaaren.
2) DNA -Polymerase aktive Zentrumstruktur. DNA -Polymerase katalysiert Nukleotiden bevorzugt mit korrekten Basen in der gegenüberliegenden Kette. Eine schlechte Paarung von Basen führt zu einer Verzerrung des Doppel -DNA -Propeller.
3) Lesetest. Die DNA -Polymerase identifiziert integrierte fehlerhafte Nukleotide und eliminiert sie aus der Tochterkette. Die Exonukleaseaktivität der Polymerase -DNA bricht die Phosphodiéster -Bindungen zwischen den Nukleotiden am 3' -Ende der neuen Kette.
DNA -Replikation in Eukaryoten
Im Gegensatz zur Replikation in Prokaryoten, deren Replikation an einer einzelnen Stelle beginnt. Anschließend werden alle Replikationsgabeln verschmolzen und bilden zwei Schwesterchromatiden, die im Zentromer verbunden sind.
Eukaryotas haben viele Arten von DNA -Polymerase, deren Namen griechische Buchstaben verwenden. DNA -Polymerase α bilden einen Komplex mit Prima. Dieser Komplex synthetisiert kurze Primer, die aus 10 RNA -Nukleotiden bestehen, gefolgt von 20 bis 30 DNA -Nukleotiden.
Dann DNA -Polymerase ε entweder δ katalysiert die Dehnung der Tochterkette aus dem Primer. DNA -Polymerase ε Es ist an der Synthese der führenden Kette beteiligt, während DNA -Polymerase δ Synthetisieren die verzögerte Kette.
DNA -Polymerase δ Verlängern Sie das linke Okazaki -Fragment, bis er den rechten RNA -Primer erreicht und ein kurzes Heben des Primers erzeugt ("Shortlap"). Im Gegensatz zu Prokaryoten, bei denen eine Polymerase -DNA den Primer eliminiert.
Als nächstes versiegelt eine DNA -Ligase die angrenzenden DNA -Fragmente. Der Abschluss der Replikation erfolgt mit der Dissoziation von Replikationsgabelproteinen.
Der DNA -Replikation in Eukaryoten und Zellzyklus
Die Replikation in Eukaryoten erfolgt in der S -Phase des Zellzyklus. Replikierte DNA -Moleküle werden während der Mitose in zwei Tochterzellen getrennt. Phasen G1 und G2 trennen die S -Phase und Mitose. Das Fortschreiten durch jede Phase des Zellzyklus wird durch Kinasen, Phosphatasen und Proteasen stark reguliert.
In der G1 -Phase des Zellzyklus verbindet der Ursprungserkennungskomplex (OCR) die Herkunftsstelle. Dies führt zu der Vereinigung von MCM-Helikas und anderen Proteinen wie Cdc6 und Cdt1, um einen Vorreplikationskomplex (PRERC) zu bilden. Las Helicase MCM schließt sich der Führungskette an.
Kann Ihnen dienen: verknüpfte GeneIn Phase S wird PRERC zu einer aktiven Replikationsstelle. OCR-, CDC6- und CDT1 -Proteine werden freigegeben, und die MCM -Helikase bewegt sich in Richtung 3 'auf 5'. Sobald die Replikation endet, wird dies im nächsten Zellzyklus neu gestartet.
Replikation der Enden von Chromosomen in Eukaryoten
Die Enden der Chromosomen werden als Telomere bezeichnet, die aus wiederholten Sequenzen im Tandem bestehen, und aus einer 3' -Region, die sich von 12 bis 16 Nukleotiden der Länge auszeichnet.
DNA -Polymerase kann das 3' -Ende der DNA -Ketten nicht replizieren. Dies liegt daran. Folglich werden Telomere in jeder Replikationsrunde verkürzt.
Telomerase -Enzym verhindert die Verkürzung der Telomere. Telomerase ist ein Enzym mit Protein- und RNA -Untereinheiten (dritte). Letzteres bindet an die wiederholten DNA -Sequenzen und ermöglicht es Telomerase, sich dem 3' -Ende des Telomers anzuschließen.
Eine Sequenz von RNA hinter der Vereinigung als Form für die Synthese einer Sequenz von sechs Nukleotiden (Polymerisation) am Ende der DNA -Kette. Die Dehnung des Telomers wird durch Telomerase -Untereinheiten katalysiert, die als Reverse Telomerase Transcriptase (Tert) bezeichnet werden.
Nach der Polymerisation findet die Translokalisierung statt, die aus der Bewegung der Telomerase zu einem neuen Ende der DNA -Kette besteht und sechs weitere Nukleotide bis zum Ende verbindet.
Die Funktionen anderer DNA -Polymerasen in den Eukaryoten
DNA -Polymerase β Es spielt eine wichtige Rolle bei der Beseitigung falscher DNA -Grundlagen, ist jedoch nicht an der DNA -Replikation beteiligt.
Viele entdeckte Polymerase-DNA gehört zur „Translesion-Replikations“ -Polymerasegruppe. Diese Polymerasen sind für die Synthese von Komplementärketten in einer beschädigten DNA -Region verantwortlich.
Es gibt verschiedene Arten von Polymerasen "Translesion-Replikation". Zum Beispiel DNA -Polymerase η Sie können sich über Timina Dímeros replizieren, die von UV -Licht erzeugt werden.
DNA -Replikation in Archaeobakterien
Die Replikation von Archaeobacteria -DNA ähnelt dem in Eukaryoten angegebenen. Dies liegt an folgenden: 1) Proteine, die an Replikation beteiligt sind, ähneln eher denen von Eukaryoten als mit denen von Prokaryoten; und 2) Obwohl es nur eine Replikationsstelle wie in Prokaryoten gibt, ähnelt seine Sequenz der Stelle des eukaryotischen Ursprungs.
Die Ähnlichkeit in der Replikation zwischen den Bögen und den Eukaryoten unterstützt die Idee, dass beide Gruppen phylogenetisch mehr miteinander verbunden sind als mit den Prokaryoten.
Verweise
- Brooker, r. J. 2018. Genetikanalyse und Prinzipien. McGraw-Hill, New York.
- Hartwell, l. H., Goldberg, m. L., Fischer, J. ZU., Haube, l. 2018. Genetik - aus Genomengenen. McGraw-Hill, New York.
- Kušić-Tišma, j. 2011. Grundlegende Aspekte der DNA -Replikation. Intech Open Access, Kroatien.
- Lewis, r., 2015. Konzepte und Anwendungen des menschlichen Genetiks. McGraw-Hill, New York.
- Pierce, b. ZU. 2005. Genetik - ein konzeptioneller Ansatz. W. H. Freeman, New York.

